Intrones: definición, función y empalme del ARN
Intrones: qué son, su función y cómo el empalme del ARN regula la expresión génica. Definición, mecanismo y su impacto en proteínas y evolución.
Un intrón es una secuencia no codificante en un gen. En términos prácticos, es cualquier tramo de nucleótidos dentro de un gen que se elimina del transcrito primario por el proceso llamado empalme de ARN para dar lugar al ARN maduro que se usará como molde para la síntesis o la función celular. El término intrón se refiere tanto a la secuencia en el ADN como a la secuencia correspondiente en los transcritos de ARN (pre-ARNm, pre-ARNr, pre-ARNt, etc.).
Estructura y señales de empalme
Los intrones suelen estar flanqueados por señales de empalme reconocibles por la maquinaria celular. En eucariotas nucleares típicas, estas señales incluyen:
- Un sitio de 5' (inicio) de intrón que frecuentemente contiene las bases conservadas GU (en el pre-ARNm típico).
- Un punto de rama (branch point) con una adenina conservada que participa en la reacción de corte y empalme.
- Una región rica en pirimidinas (tracto polipirimidínico) entre el punto de rama y el sitio 3' (final) del intrón.
- Un sitio de 3' de intrón que con frecuencia termina en las bases conservadas AG.
Estas señales no siempre son idénticas en todos los intrones, por lo que la precisión del empalme depende de la secuencia y de proteínas/HNAs auxiliares.
Tipos de intrones
- Intrones dependientes del espliceosoma (los más comunes en eucariotas): se eliminan por la maquinaria nuclear (el espliceosoma) que incluye partículas RNP pequeñas nucleares (snRNPs) y proteínas asociadas.
- Intrones autoempalmantes (grupos I y II): pueden catalizar su propio corte y empalme sin el espliceosoma; se encuentran en algunos genes de mitocondrias, cloroplastos, protozoos y bacterias.
- Intrones de ARNt y ARNr: presentan variaciones estructurales y mecanismos de procesamiento específicos.
Función y importancia biológica
Aunque a menudo se describen como "no codificantes", los intrones cumplen múltiples roles funcionales:
- Generación de diversidad proteica: a través del esplicing alternativo, diferentes combinaciones de exones pueden producir múltiples isoformas proteicas a partir de un mismo gen.
- Regulación génica: la presencia y longitud de intrones puede influir en la transcripción, en el transporte del ARNm al citoplasma y en la estabilidad del ARNm.
- Reservorio de elementos funcionales: muchos intrones contienen motivos reguladores, pequeños ARNs funcionales (como microARNs) o secuencias que afectan el empalme y la traducción.
- Facilitación de la evolución: los intrones pueden favorecer el entrecruzamiento de exones (exon shuffling) y la aparición de nuevas combinaciones funcionales.
El empalme: cuándo y cómo ocurre
El empalme del ARN ocurre después de la transcripción del ADN a un transcrito primario y antes de la traducción en la mayoría de los eucariotas. En los genes que codifican proteínas, las secuencias de ADN codificante que se conservan en el ARN maduro se llaman exones y codifican los aminoácidos del polipéptido final. El empalme preciso es crítico: un empalme erróneo puede cambiar el marco de lectura o introducir codones de parada prematuros.
La maquinaria principal que realiza el empalme en núcleos eucariotas es el espliceosoma, un complejo dinámico de snRNPs (partículas ribonucleoproteicas pequeñas nucleares) y proteínas accesorias que reconocen las señales de empalme y catalizan las reacciones de corte y unión.
Esplicing alternativo
El esplicing alternativo permite que un único gen produzca varios ARNm y proteínas diferentes según qué exones se incluyan o excluyan. Este proceso incrementa enormemente la complejidad funcional del genoma, especialmente en animales superiores, y es fundamental para el desarrollo, la diferenciación celular y la adaptación funcional.
Relevancia clínica y aplicaciones
Mutaciones que afectan sitios de empalme o proteínas del espliceosoma pueden provocar enfermedades humanas. Por ejemplo, muchas formas de beta-talasemia, distrofias y otros trastornos genéticos están asociadas a defectos de empalme. Existen además terapias basadas en oligonucleótidos antisentido que modulan el empalme (por ejemplo, en atrofia muscular espinal) y estrategias destinadas a corregir o aprovechar el empalme alternativo en medicina personalizada.
Cómo se estudian los intrones
Las técnicas modernas para investigar intrones incluyen:
- Secuenciación de ARN (RNA-Seq) para mapear transcritos y patrones de empalme.
- RT‑PCR y ensayos de minigén para analizar empalme de regiones específicas.
- Bioinformática para predecir sitios de empalme y consecuencias de variantes genéticas.
Historia del descubrimiento
El descubrimiento de los intrones y del empalme cambió profundamente la comprensión de la arquitectura génica y condujo al Premio Nobel de Fisiología o Medicina en 1993 para Phillip Sharp y Richard Roberts. El término intrón fue introducido por el bioquímico estadounidense Walter Gilbert.
- Intrones: partes de un gen que se eliminan del transcrito; no codifican aminoácidos directamente en el ARNm final, pero pueden tener funciones regulatorias.
- Exones: partes de un gen que se expresan: trozos de un gen que codifican secuencias de aminoácidos en una proteína.
En resumen, los intrones no son simplemente "bits que no funcionan": son elementos dinámicos del genoma con papel crucial en la regulación, la diversidad proteica y la evolución genética. Su estudio continúa aportando información clave sobre la biología molecular y ofrece oportunidades terapéuticas importantes.
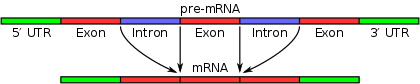
Un espliceosoma elimina los intrones de un segmento de ARNprevio transcrito (arriba). Esto se denomina "empalme". Una vez eliminados los intrones (abajo), la secuencia de ARNm maduro está lista para la traducción.
Significado biológico
Hay muchas preguntas sin respuesta sobre los intrones. No está claro si los intrones cumplen alguna función específica o si son ADN egoísta que se reproduce como un parásito.
Estudios recientes de genomas eucariotas completos han demostrado que la longitud y la densidad (intrones/genes) de los intrones varía considerablemente entre especies relacionadas. Existen cuatro o cinco tipos diferentes de intrones. Algunos intrones representan elementos genéticos móviles (transposones).
El empalme alternativo de intrones dentro de un gen permite una variedad de isoformas proteicas a partir de un único gen. Así, se pueden generar múltiples proteínas relacionadas a partir de un único gen y un único transcrito de ARNm precursor. El control del splicing alternativo del ARN se realiza mediante una compleja red de moléculas de señalización. En los seres humanos, el ~95% de los genes con más de un exón están empalmados alternativamente.
Preguntas y respuestas
P: ¿Qué es un intrón?
R: Un intrón es una secuencia no codificante en un gen que se elimina mediante el empalme de ARN para obtener el producto final de ARN de un gen.
P: ¿Qué son los exones?
R: Los exones son secuencias de ADN codificante que se unen en el ARN final tras el empalme del ARN y codifican los aminoácidos del polipéptido final.
P: ¿Dónde pueden encontrarse los intrones?
R: Los intrones pueden encontrarse en los genes de la mayoría de los organismos y de muchos virus, incluidos los que generan proteínas, ARN ribosómico (ARNr) y ARN de transferencia (ARNt).
P: ¿Cuándo tiene lugar el empalme del ARN?
R: El empalme del ARN tiene lugar después de la transcripción y antes de la traducción.
P: ¿Qué hacen los intrones?
R: Los intrones son partes de un gen que se descartan; son bits que no funcionan.
P: ¿Qué hacen los exones?
R:Los exones son partes de un gen que se expresan; codifican secuencias de aminoácidos en una proteína.
P:¿Quién descubrió los intrones?
R:El descubrimiento de los intrones condujo a la concesión del Premio Nobel de Fisiología o Medicina en 1993 a Phillip Sharp y Richard Roberts. El término intrón fue introducido por el bioquímico estadounidense Walter Gilbert.
Buscar dentro de la enciclopedia