Bioinformática: qué es la biología computacional y sus aplicaciones
Descubre la bioinformática y la biología computacional: técnicas, herramientas y aplicaciones en genómica, medicina y biotecnología para interpretar grandes datos biológicos con ordenadores.
La bioinformática o biología computacional es el estudio de grandes cantidades de información biológica. Se centra sobre todo en moléculas como el ADN. Se realiza sobre todo con la ayuda de ordenadores.
De forma más amplia, la bioinformática combina conocimientos de biología, informática, estadística y matemáticas para transformar datos experimentales en conocimiento útil. Trabaja desde la secuencia de genes hasta la estructura de proteínas y los perfiles de expresión génica, permitiendo interpretar, almacenar y visualizar grandes volúmenes de información producidos por técnicas modernas como la secuenciación masiva.
Principales tareas y métodos
- Procesamiento y gestión de datos: limpieza, formato, almacenamiento y consulta de bases de datos genómicas y proteómicas.
- Alineamiento de secuencias: comparar y alinear secuencias de ADN, ARN o proteína para identificar similitudes evolutivas o funcionales.
- Ensamblaje y anotación genómica: reconstruir genomas a partir de lecturas cortas y añadir información funcional a los genes identificados.
- Predicción de estructura y función: modelado de proteínas y predicción de dominios funcionales a partir de secuencias.
- Análisis de expresión: estudiar datos de transcriptómica (p. ej., RNA-seq) para ver qué genes se activan en distintas condiciones.
- Genómica comparada y filogenia: comparar genomas de distintas especies para entender relaciones evolutivas y conservación funcional.
- Metagenómica y ecología microbiana: analizar comunidades microbianas a partir de secuencias ambientales sin necesidad de cultivo.
- Aprendizaje automático y minería de datos: detectar patrones complejos, clasificar muestras y predecir rasgos clínicos o biológicos.
Herramientas y recursos comunes
La bioinformática emplea una mezcla de software especializado y bibliotecas de programación. Entre las herramientas y entornos más utilizados están programas de alineamiento (por ejemplo BLAST), paquetes estadísticos (como R y Bioconductor), plataformas de análisis reproducible (Galaxy), entornos de modelado molecular (PyMOL, Chimera) y lenguajes como Python para scripting y automatización. También son cruciales formatos y bases de datos estándar para facilitar el intercambio de información.
Aplicaciones prácticas
- Medicina personalizada: identificación de variantes genéticas asociadas a enfermedades y selección de tratamientos según el perfil molecular del paciente.
- Descubrimiento de fármacos: cribado virtual de compuestos, modelado de dianas y optimización de moléculas.
- Biotecnología y agricultura: mejora de cultivos, diseño de enzimas y producción de biomoléculas.
- Epidemiología y salud pública: seguimiento genómico de patógenos (por ejemplo, para rastrear brotes) y análisis de variantes.
- Investigación básica: entender la evolución, la regulación génica y las redes de interacción molecular.
Desafíos y consideraciones éticas
La bioinformática afronta retos técnicos y sociales: la gestión de volúmenes masivos de datos, la reproducibilidad de análisis, la interoperabilidad entre formatos y herramientas, y la necesidad de estándares abiertos. Además, el uso de datos genéticos plantea cuestiones de privacidad, consentimiento informado y posibles sesgos en los conjuntos de datos que pueden afectar a la equidad en salud.
Cómo formarse y empezar
Para trabajar en bioinformática es útil una base en biología molecular y en programación (especialmente Python, R y herramientas de línea de comandos en sistemas Unix/Linux). Existen cursos universitarios, másteres y numerosos cursos online (MOOCs) que cubren desde los fundamentos hasta técnicas avanzadas. Practicar con datos reales y aprender a usar repositorios y entornos reproducibles es clave para adquirir experiencia.
Tendencias futuras
El campo avanza rápidamente: la integración de datos multi-ómicos (genómica, transcriptómica, proteómica, metabolómica), la aplicación de inteligencia artificial para predicciones más precisas, y la mejora en técnicas de secuenciación y análisis en tiempo real transformarán la forma en que se investiga y aplica la biología. La bioinformática seguirá siendo esencial para convertir grandes volúmenes de datos biológicos en soluciones concretas para la ciencia, la medicina y la industria.
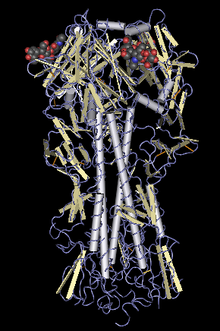
Modelo simplificado de una proteína que se encuentra en la superficie del virus de la gripe
Fundación
A medida que las especies de los seres vivos cambian con el tiempo, el ADN contenido en sus células cambia, a partir de la evolución. Si podemos extraer la información de los seres vivos actuales y compararlos entre sí, podemos ver qué seres vivos están más emparentados, de forma parecida a la comparación de dos ediciones de un libro, se puede pensar que los más parecidos son los más emparentados en el tiempo. De este modo, los biólogos pueden construir árboles genealógicos o filogenias. Al unir todos los árboles, se puede hacer un gran árbol que conecte a todos los seres vivos, lo que se llama el "Árbol de la Vida". La bioinformática es la integración de métodos matemáticos, estadísticos y computacionales para analizar datos biológicos, bioquímicos y biofísicos.
El proceso
Cualquier cosa que una célula pueda desear está almacenada en su ADN. Cuando una célula quiere construir una proteína, encuentra el trozo de ADN apropiado, hace una copia del mismo (llamada ARN) y utiliza las instrucciones de la copia para fabricar la proteína.
Las proteínas son la "maquinaria" de una célula. Pueden realizar muchas funciones, como el transporte, el soporte estructural, el movimiento y el metabolismo. Las proteínas están formadas por aminoácidos. Hay veinte aminoácidos diferentes que se utilizan para construir millones de moléculas de proteínas diferentes.
El principio de la bioinformática es que estas moléculas pueden estudiarse utilizando ordenadores para analizar las secuencias de ADN, ARN y aminoácidos a partir de las cuales se crean. Como hay tantas moléculas diferentes, la mejor manera que tenemos de entender cómo funciona todo el sistema es utilizar la bioinformática.
Informática en la bioinformática
Los químicos han desarrollado formas de entender la forma y el comportamiento de las moléculas pequeñas, utilizando el análisis matemático. Podrían utilizar ordenadores (o incluso sólo un lápiz y un papel) para estudiar estas moléculas. Además, el ADN contenido en una sola célula de un organismo es demasiado grande para ser leído por cualquier persona, y para comparar el ADN entre dos (o más) organismos, ya sea en el hermano y la hermana, o de una especie completamente diferente, se requiere comparar grandes cantidades de información para pequeñas (o grandes) diferencias. Los ordenadores son mucho más adecuados para este tipo de comparaciones, y los programadores informáticos han colaborado con los biólogos para crear bases de datos muy, muy grandes para almacenar toda la información sobre el ADN que se ha aprendido. Hoy en día, los bioquímicos intentan responder a estas preguntas sobre cada una de las células del cuerpo:
- ¿Cómo se une una determinada proteína a otra?
- ¿Qué proteínas se construirán a partir de una cadena específica de ADN?
- ¿Cómo puede utilizarse el ADN para frenar los trastornos y las enfermedades genéticas?
- ¿Cómo ha cambiado una célula a través de la evolución?
- ¿A qué enfermedades es especialmente vulnerable una persona, dados sus genes?
Páginas relacionadas
Buscar dentro de la enciclopedia