Polimorfismo de nucleótido único
Un polimorfismo de un solo nucleótido (SNP, pronunciado snip; plural snips) es una variación de la secuencia de ADN en una población. Un SNP es una diferencia de un solo nucleótido en el genoma.
Por ejemplo, los fragmentos de ADN secuenciados de dos personas, AAGCCTA a AAGCTTA, son diferentes en un solo nucleótido. En este caso decimos que hay dos alelos. Casi todos los SNP comunes tienen sólo dos alelos. Los SNP se producen con mayor frecuencia en regiones del ADN que no afectan a la supervivencia del organismo: de lo contrario, serían eliminados por la selección natural. Otros factores, como la recombinación genética y la tasa de mutación, también pueden afectar a la densidad de SNP. Existen variaciones entre las poblaciones humanas, por lo que un alelo SNP que es común en un grupo geográfico o étnico puede ser mucho más raro en otro.
Estas variaciones genéticas entre individuos (sobre todo en las partes no codificantes del genoma) se aprovechan a veces en la toma de huellas de ADN, que se utiliza en la ciencia forense. Además, estas variaciones genéticas provocan diferencias en nuestra susceptibilidad a las enfermedades. La gravedad de la enfermedad y la forma en que nuestro cuerpo responde a los tratamientos también son manifestaciones de las variaciones genéticas. Por ejemplo, una mutación de una sola base en el gen APOE (apolipoproteína E) se asocia a un mayor riesgo de padecer la enfermedad de Alzheimer.
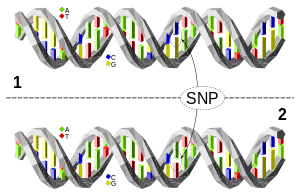
La molécula de ADN 1 difiere de la molécula de ADN 2 en un solo par de bases (un polimorfismo C/A)
Preguntas y respuestas
P: ¿Qué es un SNP?
R: Un polimorfismo de un solo nucleótido es una variación de la secuencia de ADN en una población, caracterizada por una diferencia de un solo nucleótido en el genoma.
P: ¿Qué significa SNP?
R: SNP significa polimorfismo de nucleótido único.
P: ¿Cuántos alelos tienen los SNP comunes?
R: La mayoría de los SNP comunes tienen sólo dos alelos.
P: ¿Dónde se producen con más frecuencia los SNP en el ADN?
R: Los SNP se dan con mayor frecuencia en regiones del ADN que no afectan a la supervivencia del organismo.
P: ¿Qué factores pueden afectar a la densidad de SNP?
R: Factores como la recombinación genética y la tasa de mutación también pueden afectar a la densidad de SNP.
P: ¿Cuáles son algunas aplicaciones prácticas de las variaciones genéticas entre individuos?
R: Las variaciones genéticas entre individuos (sobre todo en las partes no codificantes del genoma) se explotan a veces en la toma de huellas dactilares de ADN, que se utiliza en la ciencia forense. Estas variaciones genéticas también provocan diferencias en nuestra susceptibilidad a las enfermedades.
P: ¿Cuál es un ejemplo de variación genética vinculada a un mayor riesgo de padecer una enfermedad específica?
R: Por ejemplo, una mutación de una sola base en el gen APOE (apolipoproteína E) está asociada a un mayor riesgo de padecer la enfermedad de Alzheimer.
Buscar dentro de la enciclopedia